近日,国际综合性期刊《自然•方法》(Nature Methods)在线发表了金沙集团wwW3354CC在空间组学方面的最新研究成果。论文题为“Dependency-aware deep generative models for multitasking analysis of spatial omics data”(依赖感知的多任务深度生成模型分析空间组学数据)。金沙集团wwW3354CC为论文第一署名单位,金沙集团wwW3354CC田天教授为论文第一作者。这是我校生物信息方向研究成果首次刊登在Nature Methods期刊上。

空间组学技术是近年来生物医学研究领域的一项重大突破。不同于单细胞组学,空间组学在测序过程中同时追踪 spot 或细胞的空间位置,在组学测序数据之外提供了重要的空间信息。目前该技术为诸多研究领域提供了新的研究视角,也极大地推动了生物医疗领域的快速发展。然而如何在算法建模中高效利用空间信息数据,仍然是一个尚未解决且颇具挑战性的研究课题。对此,田天教授团队提出了一种新的对空间组学数据进行建模分析的深度学习算法,在统一的模型框架下可以进行多种下游分析任务,并且为研究其它空间组学中的生物学分析任务提供了一种新的计算框架。
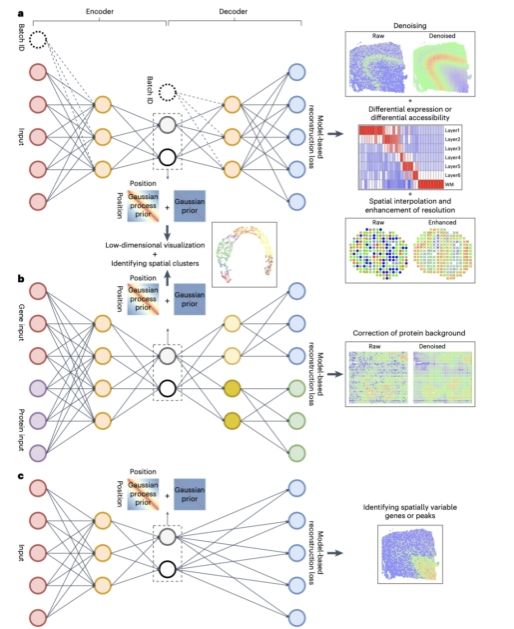
该研究基于高斯过程(Gaussian process)和变分自编码器(variational autoencoder),提出了空间依赖感知的深度生成模型 spaVAE。该模型利用高斯过程描述空间位置之间的依赖关系,得到空间转录组数据(spatially resolved transcriptomics)的统计分布,进而在统一的框架内进行降维可视化、聚类、降噪、差异表达、空间差值、提升空间分辨率等多种下游分析任务。同时,该研究进一步将spaVAE模型扩展到多种类型的空间组学数据上,例如空间染色质可及性测序(spatial ATAC-seq)和空间多组学测序(spatial CITE-seq),提出了spaPeakVAE和spaMultiVAE模型。
田天教授是国家海外优秀青年人才,共发表30余篇高水平论文,包括Nature Methods、Nature Machine Intelligence、 Nature Communications、Genome Research、EBioMedicine等。
据悉,该研究工作得到了国家海外优青项目的资助。论文地址:https://www.nature.com/articles/s41592-024-02257-y